[Python, Алгоритмы, Искусственный интеллект] Анализ результатов работы архитектуры YoloV3 на медицинских снимках (перевод)
Автор
Сообщение
news_bot ®
Стаж: 8 лет
Сообщений: 27286
Данная статья представляет собой обзор на оригинальную статью на Medium (эксперименты проводятся с изменениями некоторых условий).Область применения нейронных сетей в медицине бурно развивается. В этой области решаются задачи, которые облегчают работу врачей. В частности, одной из востребованных задач в этой области является детекция объектов на медицинских снимках (это когда на картинку накладывается прямоугольник, который ограничивает область, в которой предположительно есть некоторый объект). Пример такого изображения представлен ниже.
https://github.com/ultralytics/yolov3https://github...tics/yolov3Можно заметить, что прямоугольники подписаны какими-то словами и числами. На картинке это person и tie. Рядом с этими словами написаны числа (у человека слева это person с 0.59, tie - 0.62). Эти слова образуют виды объектов (например, машина, человек, кот, мяч и т.д.), которые нужно распознать, а числа, записанные рядом с этими словами, есть вероятность того, что данный объект принадлежит этому классу. (Опять же у человека справа, стоит "person 0.59". Это значит, что в выделенном прямоугольнике есть объект класса person - человек - с вероятностью 0.59). И да, число - вероятность объекта в данном прямоугольника, принимает значения от 0 до 1. ЗадачаКак уже говорилось, в медицине есть задача по распознаванию объектов, которые могут сигнализировать о наличии патологии и пациента. В данной статье, предлагается решить задачу по распознаванию очагов, сигнализирующих пневмонию у пациента. Пневмония является одной из распространенных болезней, которое представляет собой воспалительное заболевание легких. По официальным данным, именно она является одной из опасных инфекционных заболеваний за последние 20 лет. В 2019-м эти болезни оказались четвертой причиной смертности в мире (от них скончались 2,6 млн человек). Обычно пневмония проявляется в виде областей повышенной непрозрачности на снимках рентгенограммы. Однако диагностика рентгенограмм затруднена из-за ряда причин, связанных с состоянием легких. И иногда даже опытному специалисту бывает сложно поставить диагноз. Поэтому на помощь врачам приходят методы машинного обучения, которые помогают выявить сложные зависимости между признаками в данных и выдать некоторый результат, который может стать решающим при постановке диагноза пациенту.В связи с этим, возникает потребность в написании нейронных сетей, которые основаны на совершенно новой архитектуре (то есть придумать что-то новое) или которые основаны на уже существующей архитектуры путем проведения экспериментов, которые помогают выявлять достоинства и недостатки архитектуры (то есть сделать модификацию существующей). Решать эту задачу мы будем с использованием нейронной сети.Модель В качестве такой сети возьмем архитектуру YOLOv3. Почему именно она? Да, просто захотели =) Более подробно про эту архитектуру можно почитать на официальном сайте и Хабре. YOLOv3 представляет собой нейронную сеть, основанную на архитектуре YOLO (You Only Look Once). Она примечательна тем, что CNN (Convolutional Neural Network) применяется один раз ко всему изображению сразу (отсюда и название). YOLOv3 состоит из 106-ти свёрточных слоев. Стоит отметить, что у YOLOv3 есть несколько слоев (их 3), которые предназначены для детекции объектов разного размера. На картинке ниже представлена архитектура YOLOv3: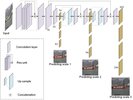
https://www.researchgate.net/figure/The-framework-o...ig2_335228064При использовании YOLO изображение делится на сетку с ячейками размером 13 х 13. Для чего нужны эти ячейки? Дело в том, что каждая такая ячейка прогнозирует количество bounding box'ов (или ограничивающих прямоугольников) и вероятность того, что в данной области находится некоторый объект. Эта вероятность (точнее, число) называется confidence value (доверительное значение). И получается, что если в некоторой области объекта нет, то его доверительное значение маленькое (точнее, этого мы хотим достичь). Ниже представлена схема работы YOLOv3.
https://medium.com/nerd-for-tech/a-real-time-object...8941a20b445Также примечательно, что YOLO использует, так называемые anchor boxes (якорные рамки). Подробнее о них написано в статье на Medium. Это достаточно сложная для понимания(лично для автора этой статьи) концепция. Нам важно лишь то, что anchor boxes (якорные рамки) используются для прогнозирования bounding box'ов и рассчитаны они с помощью датасета COCO с использованием кластеризации k-средних. Чтобы более подробно познакомиться с YOLOv3 подойдет вот эта статья. ДанныеС задачей определились, с моделью определились. Что еще надо? Правильно, данные. Данные берутся из платформы Kaggle, в которой проводились соревнования по детекции пневмонии. Вот данные.Изучим эти данные более подробно. Нам понадобятся изображения из файлов stage_2_train_images.zip и stage_2_test_images.zip. Данные, которые давались на соревновании, представляют собой набор снимков рентгенограммы грудной клетки. В датасете (а именно так называются набор данных) содержатся 26684 рентгеновских снимков разных пациентов. Данные снимки представляют собой изображения в формате DICOM в разрешении 1024 х 1024. Пример изображения представлен ниже.
ClassTargetPatientsLung Opacity19555No Lung Opacity / Not Normal011821Normal08851Так как изображения находятся в формате DICOM. То мы преобразуем эти изображения в формат JPG с помощью следующей функции.
import pydicom as dicom
import os
from tqdm import tqdm
import numpy as np
import cv2
import pandas as pd
перевод dicom в jpg
def dicom_to_jpg(source_folder,destination_folder,labels):
images_path = os.listdir(source_folder)
image_dirs_label = {'image_dir':[],'Target':[]}
for n, image in tqdm(enumerate(images_path)):
ds = dicom.dcmread(os.path.join(source_folder, image))
pixel_array_numpy = ds.pixel_array
image = image.replace('.dcm', '.jpg')
cv2.imwrite(os.path.join(destination_folder, image), pixel_array_numpy)
image_dirs_label['image_dir'].append(os.path.join(destination_folder, image))
image_dirs_label['Target'].append(train_labels[train_labels.patientId== image.split('.')[0]].Target.values[0])
print('{} dicom files converted to jpg!'.format(len(images_path)))
return pd.DataFrame(image_dirs_label)
Выделяются 3 класса, которые представляют для интерес: Normal — 0, No Lung Opacity / Not Normal — 0, Lung Opacity — 1. Классы Class, целевые признаки Target и количество изображений Patients, соответствующего класса, представлены в таблице выше. И картинка ниже показывает изображения каждого класса.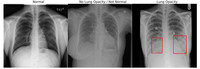
Для нас особый интерес представляют классы, сигнализирующие пневмонию (positive или на картинке выше Lung Opacity). И соотношение этого класса к классу изображений здоровых пациентов (negative) равно примерно 1:4 (ниже есть диаграмма, иллюстрирующая данное соотгношение). 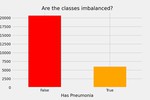
Дисбаланс классовДисбаланс классовТо есть классы несбалансированы (изображений одного класса больше, чем изображений другого). Поэтому для достижения относительного равенства между классами был использован прием увеличения числа изображений первого класса (positive) из уже имеющихся путем их преобразований — аугментация. Аугментация была реализована с помощью библиотеки Albumentations. Ниже представлен код для совершения аугментации.
import albumentations as A
import pandas as pd
import cv2
import os
transformer
transform = A.Compose([
A.RandomRotate90(),
A.Flip(),
A.Transpose(),
A.OneOf([
A.IAAAdditiveGaussianNoise(),
A.GaussNoise(),
], p=0.2),
A.OneOf([
A.MotionBlur(p=.2),
A.MedianBlur(blur_limit=3, p=0.1),
A.Blur(blur_limit=3, p=0.1),
], p=0.2),
A.ShiftScaleRotate(shift_limit=0.0625, scale_limit=0.2, rotate_limit=45, p=0.2),
A.OneOf([
A.OpticalDistortion(p=0.3),
A.GridDistortion(p=.1),
A.IAAPiecewiseAffine(p=0.3),
], p=0.2),
A.OneOf([
A.CLAHE(clip_limit=2),
A.IAASharpen(),
A.IAAEmboss(),
A.RandomBrightnessContrast(), ], p=0.3),
A.HueSaturationValue(p=0.3),
])
Реализация моделиДанный раздел будет обновляться в будущем(ибо есть технические "подводные камни", о которых не рассказано, но о которых стоит рассказать)Теперь датасет мы "расширили". Преобразованные изображения и исходные файлы в форматах JPG и DICOM будем анализировать с использованием архитектуры YOLOv3 с основой (backbone'ом) DarkNet. Подробнее про DarkNet можно почитать здесь. Затем основа архитектуры YOLOv3 (в данном случае Darknet) заменяется на обученную классификационную модель CheXNet. CheXNet представляет собой 121-слойную свёрточую нейронную сеть, которая определяет области легких, сигнализирующих о пневмонии. Рекомендуется прочитать эту научную работупро CheXNet. Эта модель обучена на классификацию 14 классов, поэтому так как мы решаем задачу бинарной классификации, то последние слои CheXNet необходимо установить на классификацию 2-х классов (negative — пневмонии нет и positive — пневмония есть). И реализовать в коде данную модель можно с помощью библиотеки TensorFlow, в которой есть готовая заготовка DenseNet121. Реализация этой модели представлено ниже.
# Для CheXNet устанавлиются веса classifier_weights.hdf5, которые можно скачать отсюда
https://drive.google.com/file/d/1Bd50DpRWorGMDuEZ3-VHgndpJZwUGTAr/view
from absl import flags
from absl.flags import FLAGS
import numpy as np
import tensorflow as tf
from tensorflow.keras import Model
from tensorflow.keras.applications import DenseNet121
from tensorflow.keras.layers import (
Add,
Concatenate,
Conv2D,
Input,
Lambda,
LeakyReLU,
MaxPool2D,
UpSampling2D,
ZeroPadding2D,
BatchNormalization,
Dense
)
def base_model(chexnet_weights=None,size=None):
dense_net_121 = DenseNet121(input_shape = [size,size,3], include_top = False,pooling = 'avg')
base_model_output = Dense(units = 14, activation = 'relu')(dense_net_121.output)
base_model = Model(inputs = dense_net_121.input,outputs = base_model_output)
output_layer = Dense(1, activation = 'sigmoid')(base_model.layers[-2].output)
model = Model(inputs = base_model.inputs, outputs = output_layer)
if chexnet_weights:
model.load_weights(chexnet_weights)
final_base_model = Model(inputs = model.inputs, outputs = model.layers[-3].output)
return final_base_model
def ChexNet(name=None, chexnet_weights='PATH_TO_WEIGTHS/classifier_weights.hdf5',size=None):
chexnet = base_model(chexnet_weights = chexnet_weights, size = size)
back_bone = Model(inputs = chexnet.inputs, outputs=(chexnet.get_layer('pool3_conv').output,
chexnet.get_layer('pool4_conv').output,
chexnet.output),name=name)
return back_bone
Теперь посмотрим на количество параметров каждой модели:ModelTotal paramsTrainable paramsNon-trainable paramsDarkNet 61576342 61523734 52608CheXNet 27993206 27892662 100544Видим, что параметров у архитектуры с классификационной моделью CheXNet почти в 2 раза меньше параметров, чем у архитектуры с классификационной моделью DarkNet. Это делает первую модель более быстрой в обучении и по этой причине дальнейшая работа будет производиться именно с CheXNet.ОбучениеПолученная архитектура нейронной сети YOLOv3 с основой CheXNet обучается на преобразованных данных(над которыми был совершен процесс аугментации). Стоит отметить то, что мы сначала обучаем (1 эпоху) на всех классах изображений (positive и negative), а затем на изображениях, в которых есть пневмония (класса positive). Это делается потому что в YOLOv3 изображение 416 х 416 делится на сетку 13 х 13 (416 / 32 = 13). И прогноз делается для каждой ячейки сетки 13 х 13. И если количество anchor box'ов равно 3, тогда каждая такая ячейка сетки 13 х 13 связана с 3-мя anchor box'ами. То есть размерность будет 13 х 13 х 3 = 507 (всего будет столько предсказаний). Получается, что для одного изображения мы делаем 507 предсказаний. И даже если изображение относится к классу positive (пневмония есть) и в нем есть 2 области непрозрачности (помутнения), то будет 2 положительных предсказания и 507-2=505 отрицательных предсказаний. Как видно, число отрицательных предсказаний намного больше. Поэтому если мы снова добавим отрицательные изображения, это сделает нашу модель "предвзятой" по отношению к отрицательному классу. Для начала, мы делаем ImageDataGenerator для обучения модели. Это связано с тем, что набор данных достаточно большой (и он не поместится в оперативную память), а данный инструмент позволяет нам облегчить чтение изображений во время обучения модели.
# true_augmented_labels - это DataFrame, который содержит информацию
о всех изображениях (и о изначальных, и аугментированных(преобразованных)
datagen=ImageDataGenerator(
rescale = 1. / 255.,
validation_split = 0.20)
train_generator = datagen.flow_from_dataframe(
dataframe = true_augmented_labels,
x_col = "image_dir",
y_col = "Target",
subset = "training",
batch_size = 4,
seed = 42,
shuffle = True,
class_mode = "binary",
target_size = (416, 416))
valid_generator = datagen.flow_from_dataframe(
dataframe = true_augmented_labels,
x_col = "image_dir",
y_col = "Target",
subset = "validation",
batch_size = 4,
seed = 42,
shuffle = True,
class_mode = "binary",
target_size = (416, 416))
Затем мы обучаем нашу модель на всех классах изображений (и positive, и negative), заранее замораживая последние слои модели.
# веса brucechou1983_CheXNet_Keras_0.3.0_weights.h5 и classifier_weights.hdf5
можно скачать отсюда https://www.kaggle.com/theewok/chexnet-keras-weights/version/1
и отсюда https://github.com/junaidnasirkhan/Replacing-YoloV3-Backbone-with-ChexNet-for-Pneumonia-Detection
dense_net_121 = DenseNet121(input_shape = [416,416] + [3], include_top = False, pooling = 'avg')
base_model_output = Dense(units = 14, activation = 'relu')(dense_net_121.output)
base_model = Model(inputs = dense_net_121.input, outputs = base_model_output)
загрузка "тренированных" весов
base_model.load_weights('brucechou1983_CheXNet_Keras_0.3.0_weights.h5')
заморозка последних слоев модели
for layer in base_model.layers[:10]:
layer.trainable = False
устанавлием последние слои модели на бинарную классификацию
output_layer = Dense(1, activation = 'sigmoid')(base_model.layers[-2].output)
model = Model(inputs = base_model.inputs, outputs = output_layer)
model.compile(optimizer = 'adam', loss = 'binary_crossentropy', metrics = ['accuracy', f1_m])
checkpoint = ModelCheckpoint(filepath = 'classifier_weights.hdf5', monitor = 'val_accuracy', verbose = 0, save_best_only = True, save_weights_only = True, mode = 'auto')
log_dir = "classifier_logs/" + datetime.datetime.now().strftime("%Y%m%d-%H%M%S")
tensorboard = TensorBoard(log_dir = log_dir, histogram_freq = 1, write_graph = True, write_grads = True)
callback_list = [checkpoint, tensorboard]
обучаем модель
model.fit(train_generator,
validation_data = valid_generator,
epochs = 1, # в оригинальной статье стоит 3
steps_per_epoch = len(train_generator),
callbacks = callback_list)
Затем нам надо написать функцию обучения на positive изображениях (причина описана выше). Она представлена ниже
# Для обучения модели были созданы файлы rsna_train_pos.tfrecord и rsna_val_pos.tfrecord
Классы изображений записываются в формате .names (в нашем случае)
это классы "opacity" и "no_opacity"
model = train(dataset = 'PATH_TO_TFRECORD/rsna_train_pos.tfrecord',
val_dataset = 'PATH_TO_TFRECORD/rsna_val_pos.tfrecord',
backbone = 'chexnet',
classes = 'PATH_TO_CLASSES/RSNA_VOC.names',
size = 416,
epochs = 30,
batch_size = 16, learning_rate = 1e-4,
num_classes = 1)
После обучения веса модели бинарной классификации сохраняются в виде файла формата hdf5. Результаты обученияНиже представлен результат обучения данной архитектуры (YOLOv3 с классификационной моделью CheXNet). С параметрами learning_rate = 1e-4, epoch = 20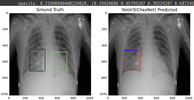
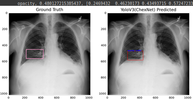
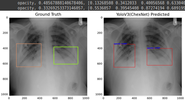
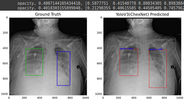
Посмотрим на loss'ы
Аналогично для learning_rate = 1e-4, epochs = 30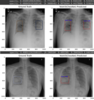
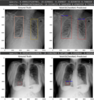
Посмотрим на loss'ы
Выводы
- Над исходными данными был совершен процесс аугментации с целью увеличения количества данных.
- Анализ количество обучаемых параметров моделей CheXNet и DarkNet показал, что таких параметров меньше у модели CheXNet, что делает ее обучение быстрым по сравнению с обучением модели DarkNet.
- Архитектура с классификационной моделью CheXNet была обучена 1 эпоху на изображениях всех классов, а затем 20 эпох и 30 эпох на изображениях, содержащих признаки пневмонии.
- Эксперименты показали, что с увеличением числа epoch, растет и точность предсказаний модели.
ПерспективыРассматривается возможность улучшения показателей архитектуры. Этого можно достичь путем:
- обучения с изменением параметров (увеличения количества эпох, значения learning_rate)
- обучения модели с использованием другого датасета
- модель CheXNet можно заменить другую классификационную модель
Ссылки
- На оригинальную статью на Medium
- На мой GitHub
===========
Источник:
habr.com
===========
===========
Автор оригинала: Junaid Khan
===========Похожие новости:
- [Информационная безопасность, Python, Программирование, Алгоритмы] Реализация алгоритмов хеширования семейства SHA-2
- [Искусственный интеллект, Мультикоптеры] Автономный военный дрон впервые самостоятельно атаковал человека
- [Python] Сокрытые драгоценности Python (перевод)
- [Ruby, Python, Программирование, Будущее здесь] Почему за интерпретируемыми языками будущее
- [Машинное обучение, Научно-популярное, Искусственный интеллект, IT-компании, Serverless] Российские ученые совместно с «Яндексом» создали систему мониторинга сельхозрастений с помощью нейросети
- [Машинное обучение, Научно-популярное, Искусственный интеллект, Игры и игровые приставки] Как StarCraft II может помочь экологам в изучении жизни на Земле (перевод)
- [JavaScript, Программирование, Алгоритмы, Функциональное программирование] Решаем вопрос сортировки в JavaScript раз и навсегда
- [Open source, Python, Алгоритмы, Машинное обучение, Искусственный интеллект] Прогнозирование временных рядов с помощью AutoML
- [Занимательные задачки, Python, Алгоритмы, Математика] В аквариуме: вычислительная генетика на Python и Mathcad (часть 1)
- [Разработка веб-сайтов, Python, JavaScript, Программирование] Как создавать предметы генеративного искусства с помощью L-систем на языке Python (перевод)
Теги для поиска: #_python, #_algoritmy (Алгоритмы), #_iskusstvennyj_intellekt (Искусственный интеллект), #_iskusstvennyj_intellekt (искусственный интеллект), #_algoritmy (алгоритмы), #_meditsina (медицина), #_kaggle, #_python, #_algoritmy (
Алгоритмы
), #_iskusstvennyj_intellekt (
Искусственный интеллект
)
Вы не можете начинать темы
Вы не можете отвечать на сообщения
Вы не можете редактировать свои сообщения
Вы не можете удалять свои сообщения
Вы не можете голосовать в опросах
Вы не можете прикреплять файлы к сообщениям
Вы не можете скачивать файлы
Текущее время: 11-Фев 12:11
Часовой пояс: UTC + 5
| Автор | Сообщение |
|---|---|
|
news_bot ®
Стаж: 8 лет |
|
|
Данная статья представляет собой обзор на оригинальную статью на Medium (эксперименты проводятся с изменениями некоторых условий).Область применения нейронных сетей в медицине бурно развивается. В этой области решаются задачи, которые облегчают работу врачей. В частности, одной из востребованных задач в этой области является детекция объектов на медицинских снимках (это когда на картинку накладывается прямоугольник, который ограничивает область, в которой предположительно есть некоторый объект). Пример такого изображения представлен ниже.  https://github.com/ultralytics/yolov3https://github...tics/yolov3Можно заметить, что прямоугольники подписаны какими-то словами и числами. На картинке это person и tie. Рядом с этими словами написаны числа (у человека слева это person с 0.59, tie - 0.62). Эти слова образуют виды объектов (например, машина, человек, кот, мяч и т.д.), которые нужно распознать, а числа, записанные рядом с этими словами, есть вероятность того, что данный объект принадлежит этому классу. (Опять же у человека справа, стоит "person 0.59". Это значит, что в выделенном прямоугольнике есть объект класса person - человек - с вероятностью 0.59). И да, число - вероятность объекта в данном прямоугольника, принимает значения от 0 до 1. ЗадачаКак уже говорилось, в медицине есть задача по распознаванию объектов, которые могут сигнализировать о наличии патологии и пациента. В данной статье, предлагается решить задачу по распознаванию очагов, сигнализирующих пневмонию у пациента. Пневмония является одной из распространенных болезней, которое представляет собой воспалительное заболевание легких. По официальным данным, именно она является одной из опасных инфекционных заболеваний за последние 20 лет. В 2019-м эти болезни оказались четвертой причиной смертности в мире (от них скончались 2,6 млн человек). Обычно пневмония проявляется в виде областей повышенной непрозрачности на снимках рентгенограммы. Однако диагностика рентгенограмм затруднена из-за ряда причин, связанных с состоянием легких. И иногда даже опытному специалисту бывает сложно поставить диагноз. Поэтому на помощь врачам приходят методы машинного обучения, которые помогают выявить сложные зависимости между признаками в данных и выдать некоторый результат, который может стать решающим при постановке диагноза пациенту.В связи с этим, возникает потребность в написании нейронных сетей, которые основаны на совершенно новой архитектуре (то есть придумать что-то новое) или которые основаны на уже существующей архитектуры путем проведения экспериментов, которые помогают выявлять достоинства и недостатки архитектуры (то есть сделать модификацию существующей). Решать эту задачу мы будем с использованием нейронной сети.Модель В качестве такой сети возьмем архитектуру YOLOv3. Почему именно она? Да, просто захотели =) Более подробно про эту архитектуру можно почитать на официальном сайте и Хабре. YOLOv3 представляет собой нейронную сеть, основанную на архитектуре YOLO (You Only Look Once). Она примечательна тем, что CNN (Convolutional Neural Network) применяется один раз ко всему изображению сразу (отсюда и название). YOLOv3 состоит из 106-ти свёрточных слоев. Стоит отметить, что у YOLOv3 есть несколько слоев (их 3), которые предназначены для детекции объектов разного размера. На картинке ниже представлена архитектура YOLOv3:  https://www.researchgate.net/figure/The-framework-o...ig2_335228064При использовании YOLO изображение делится на сетку с ячейками размером 13 х 13. Для чего нужны эти ячейки? Дело в том, что каждая такая ячейка прогнозирует количество bounding box'ов (или ограничивающих прямоугольников) и вероятность того, что в данной области находится некоторый объект. Эта вероятность (точнее, число) называется confidence value (доверительное значение). И получается, что если в некоторой области объекта нет, то его доверительное значение маленькое (точнее, этого мы хотим достичь). Ниже представлена схема работы YOLOv3.  https://medium.com/nerd-for-tech/a-real-time-object...8941a20b445Также примечательно, что YOLO использует, так называемые anchor boxes (якорные рамки). Подробнее о них написано в статье на Medium. Это достаточно сложная для понимания(лично для автора этой статьи) концепция. Нам важно лишь то, что anchor boxes (якорные рамки) используются для прогнозирования bounding box'ов и рассчитаны они с помощью датасета COCO с использованием кластеризации k-средних. Чтобы более подробно познакомиться с YOLOv3 подойдет вот эта статья. ДанныеС задачей определились, с моделью определились. Что еще надо? Правильно, данные. Данные берутся из платформы Kaggle, в которой проводились соревнования по детекции пневмонии. Вот данные.Изучим эти данные более подробно. Нам понадобятся изображения из файлов stage_2_train_images.zip и stage_2_test_images.zip. Данные, которые давались на соревновании, представляют собой набор снимков рентгенограммы грудной клетки. В датасете (а именно так называются набор данных) содержатся 26684 рентгеновских снимков разных пациентов. Данные снимки представляют собой изображения в формате DICOM в разрешении 1024 х 1024. Пример изображения представлен ниже.  ClassTargetPatientsLung Opacity19555No Lung Opacity / Not Normal011821Normal08851Так как изображения находятся в формате DICOM. То мы преобразуем эти изображения в формат JPG с помощью следующей функции. import pydicom as dicom
import os from tqdm import tqdm import numpy as np import cv2 import pandas as pd перевод dicom в jpg def dicom_to_jpg(source_folder,destination_folder,labels): images_path = os.listdir(source_folder) image_dirs_label = {'image_dir':[],'Target':[]} for n, image in tqdm(enumerate(images_path)): ds = dicom.dcmread(os.path.join(source_folder, image)) pixel_array_numpy = ds.pixel_array image = image.replace('.dcm', '.jpg') cv2.imwrite(os.path.join(destination_folder, image), pixel_array_numpy) image_dirs_label['image_dir'].append(os.path.join(destination_folder, image)) image_dirs_label['Target'].append(train_labels[train_labels.patientId== image.split('.')[0]].Target.values[0]) print('{} dicom files converted to jpg!'.format(len(images_path))) return pd.DataFrame(image_dirs_label)  Для нас особый интерес представляют классы, сигнализирующие пневмонию (positive или на картинке выше Lung Opacity). И соотношение этого класса к классу изображений здоровых пациентов (negative) равно примерно 1:4 (ниже есть диаграмма, иллюстрирующая данное соотгношение).  Дисбаланс классовДисбаланс классовТо есть классы несбалансированы (изображений одного класса больше, чем изображений другого). Поэтому для достижения относительного равенства между классами был использован прием увеличения числа изображений первого класса (positive) из уже имеющихся путем их преобразований — аугментация. Аугментация была реализована с помощью библиотеки Albumentations. Ниже представлен код для совершения аугментации. import albumentations as A
import pandas as pd import cv2 import os transformer transform = A.Compose([ A.RandomRotate90(), A.Flip(), A.Transpose(), A.OneOf([ A.IAAAdditiveGaussianNoise(), A.GaussNoise(), ], p=0.2), A.OneOf([ A.MotionBlur(p=.2), A.MedianBlur(blur_limit=3, p=0.1), A.Blur(blur_limit=3, p=0.1), ], p=0.2), A.ShiftScaleRotate(shift_limit=0.0625, scale_limit=0.2, rotate_limit=45, p=0.2), A.OneOf([ A.OpticalDistortion(p=0.3), A.GridDistortion(p=.1), A.IAAPiecewiseAffine(p=0.3), ], p=0.2), A.OneOf([ A.CLAHE(clip_limit=2), A.IAASharpen(), A.IAAEmboss(), A.RandomBrightnessContrast(), ], p=0.3), A.HueSaturationValue(p=0.3), ]) # Для CheXNet устанавлиются веса classifier_weights.hdf5, которые можно скачать отсюда
https://drive.google.com/file/d/1Bd50DpRWorGMDuEZ3-VHgndpJZwUGTAr/view from absl import flags from absl.flags import FLAGS import numpy as np import tensorflow as tf from tensorflow.keras import Model from tensorflow.keras.applications import DenseNet121 from tensorflow.keras.layers import ( Add, Concatenate, Conv2D, Input, Lambda, LeakyReLU, MaxPool2D, UpSampling2D, ZeroPadding2D, BatchNormalization, Dense ) def base_model(chexnet_weights=None,size=None): dense_net_121 = DenseNet121(input_shape = [size,size,3], include_top = False,pooling = 'avg') base_model_output = Dense(units = 14, activation = 'relu')(dense_net_121.output) base_model = Model(inputs = dense_net_121.input,outputs = base_model_output) output_layer = Dense(1, activation = 'sigmoid')(base_model.layers[-2].output) model = Model(inputs = base_model.inputs, outputs = output_layer) if chexnet_weights: model.load_weights(chexnet_weights) final_base_model = Model(inputs = model.inputs, outputs = model.layers[-3].output) return final_base_model def ChexNet(name=None, chexnet_weights='PATH_TO_WEIGTHS/classifier_weights.hdf5',size=None): chexnet = base_model(chexnet_weights = chexnet_weights, size = size) back_bone = Model(inputs = chexnet.inputs, outputs=(chexnet.get_layer('pool3_conv').output, chexnet.get_layer('pool4_conv').output, chexnet.output),name=name) return back_bone # true_augmented_labels - это DataFrame, который содержит информацию
о всех изображениях (и о изначальных, и аугментированных(преобразованных) datagen=ImageDataGenerator( rescale = 1. / 255., validation_split = 0.20) train_generator = datagen.flow_from_dataframe( dataframe = true_augmented_labels, x_col = "image_dir", y_col = "Target", subset = "training", batch_size = 4, seed = 42, shuffle = True, class_mode = "binary", target_size = (416, 416)) valid_generator = datagen.flow_from_dataframe( dataframe = true_augmented_labels, x_col = "image_dir", y_col = "Target", subset = "validation", batch_size = 4, seed = 42, shuffle = True, class_mode = "binary", target_size = (416, 416)) # веса brucechou1983_CheXNet_Keras_0.3.0_weights.h5 и classifier_weights.hdf5
можно скачать отсюда https://www.kaggle.com/theewok/chexnet-keras-weights/version/1 и отсюда https://github.com/junaidnasirkhan/Replacing-YoloV3-Backbone-with-ChexNet-for-Pneumonia-Detection dense_net_121 = DenseNet121(input_shape = [416,416] + [3], include_top = False, pooling = 'avg') base_model_output = Dense(units = 14, activation = 'relu')(dense_net_121.output) base_model = Model(inputs = dense_net_121.input, outputs = base_model_output) загрузка "тренированных" весов base_model.load_weights('brucechou1983_CheXNet_Keras_0.3.0_weights.h5') заморозка последних слоев модели for layer in base_model.layers[:10]: layer.trainable = False устанавлием последние слои модели на бинарную классификацию output_layer = Dense(1, activation = 'sigmoid')(base_model.layers[-2].output) model = Model(inputs = base_model.inputs, outputs = output_layer) model.compile(optimizer = 'adam', loss = 'binary_crossentropy', metrics = ['accuracy', f1_m]) checkpoint = ModelCheckpoint(filepath = 'classifier_weights.hdf5', monitor = 'val_accuracy', verbose = 0, save_best_only = True, save_weights_only = True, mode = 'auto') log_dir = "classifier_logs/" + datetime.datetime.now().strftime("%Y%m%d-%H%M%S") tensorboard = TensorBoard(log_dir = log_dir, histogram_freq = 1, write_graph = True, write_grads = True) callback_list = [checkpoint, tensorboard] обучаем модель model.fit(train_generator, validation_data = valid_generator, epochs = 1, # в оригинальной статье стоит 3 steps_per_epoch = len(train_generator), callbacks = callback_list) # Для обучения модели были созданы файлы rsna_train_pos.tfrecord и rsna_val_pos.tfrecord
Классы изображений записываются в формате .names (в нашем случае) это классы "opacity" и "no_opacity" model = train(dataset = 'PATH_TO_TFRECORD/rsna_train_pos.tfrecord', val_dataset = 'PATH_TO_TFRECORD/rsna_val_pos.tfrecord', backbone = 'chexnet', classes = 'PATH_TO_CLASSES/RSNA_VOC.names', size = 416, epochs = 30, batch_size = 16, learning_rate = 1e-4, num_classes = 1)     Посмотрим на loss'ы  Аналогично для learning_rate = 1e-4, epochs = 30   Посмотрим на loss'ы  Выводы
=========== Источник: habr.com =========== =========== Автор оригинала: Junaid Khan ===========Похожие новости:
Алгоритмы ), #_iskusstvennyj_intellekt ( Искусственный интеллект ) |
|
Вы не можете начинать темы
Вы не можете отвечать на сообщения
Вы не можете редактировать свои сообщения
Вы не можете удалять свои сообщения
Вы не можете голосовать в опросах
Вы не можете прикреплять файлы к сообщениям
Вы не можете скачивать файлы
Вы не можете отвечать на сообщения
Вы не можете редактировать свои сообщения
Вы не можете удалять свои сообщения
Вы не можете голосовать в опросах
Вы не можете прикреплять файлы к сообщениям
Вы не можете скачивать файлы
Текущее время: 11-Фев 12:11
Часовой пояс: UTC + 5
