[Машинное обучение, Научно-популярное, Искусственный интеллект, Здоровье] ИИ ищет в микробах соединения для новых лекарств
Автор
Сообщение
news_bot ®
Стаж: 8 лет
Сообщений: 27286
Исследователи Университета Карнеги — Меллона обучили нейросеть искать в микробах метаболиты (нерибосомные пептиды) и оценивать их потенциальную пользу для разработки лекарств. 
В процессе жизнедеятельности бактерии и грибки синтезируют вторичные метаболиты, в том числе нерибосомные пептиды (NRP). Эти пептиды учёные используют для производства антибиотиков, противоопухолевых препаратов и иммунодепрессантов. Например, один из самых мощных антибиотиков ванкомицин исследователи нашли в продуктах метаболизма бактерии Amycolatopsis orientalis. В последние годы исследователи отходят от поиска NRP в микробах. Процесс поиска трудоёмкий и требует много времени на оценку обнаруженных соединений. Не все пептиды пригодны для производства лекарств. Поэтому специалисты Университета Карнеги-Меллона создали алгоритм, упрощающий процессы поиска и анализа. Исследователи назвали платформу NRPminer и выложили на GitHub. Для исследования учёные взяли метаболиты штаммов микробов семейств Actinobacteria, Xenorhabdus, Photorhabdus, Staphylococcus и почвенных бактерий Bacillus, Pseudomonas, Buttiauxella и Rahnella. Всего отобрали 246 штаммов. В ходе анализа NRPminer идентифицировала биосинтетические генные кластеры пептидов (BGC), используя пайплайн antiSMASH16. (пункт “а” на рисунке ниже) Система предсказывала вариации аминокислот с помощью веб-сервера NRPSpredictor2. (b) Далее она рассмотрела сформированные NRPS последовательности нуклеотидов. (с) NRPminer отфильтровала набор основных последовательностей NRP на основе оценки специфичности аминокислот и выбрала варианты с самыми высокими оценками. (d) Платформа нашла каждый BGC и включила их в последовательности NRP. (e) Она создала базу данных потенциальных структур NPR (f), после чего искала подходящие модификации и вычислила их статистическую значимость. (g) Компонент NRPS выделил и расширил статистически значимые показатели с использованием метода спектральных сетей. (h и i) Обнаруженные вариации NRP алгоритм сопоставил с общедоступным набором спектральных данных геномов RefSeq82 и онлайн-базой данных геномов GOLD. 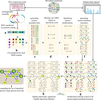
В итоге NRPminer обнаружил 13 известных семейств NRP (55 вариаций) и 4 неизвестных семейства с 25 вариациями. Исследователи уже изучили неизвестные семейства и выделили у двух антипаразитарную активность. Кроме того, анализ показал наличие 121 известной вариации NRP у микробов, к которым исследователи ранее не относили эти пептиды. Алгоритм определяет только 1 % продуктов метаболизма микробов. Остальные 99 % включают в себя первичные метаболиты (аминокислоты), другие вторичные метаболиты (RiPP, поликетиды, липиды, терпены), молекулы из окружающей среды и элементы с низкой интенсивностью, которые сложно обнаружить. 
Спектральные сети для девяти известных и трёх ранее неизвестных семейств NRP. Каждый узел на изображении обозначает спектр NRP. Узел окрашен, если спектр обладает статистической значимостью. Известные NRP окрашены голубым. Светло-зелёным отмечены неизвестные NRP. Спектры ранее неизвестных семейств NRP окрашены сиреневым.Исследователи отмечают необходимость дальнейшего пополнения базы данных и повышения чувствительности NRPminer. В ходе анализа авторы столкнулись с проблемой наличия в результатах масс-спектрометрии спектров из окружающей среды. Например, большинство спектров микробиома кожи человека соответствует косметических средствам (крема, дезодоранты). Учёные заявили, что появление более чувствительных методом спектрометрии решит эту проблему. Согласно заявлению авторов исследования, NRPminer — первый генетический инструмент, эффективно фильтрующий основные NRP на основе показателей специфичности без потери чувствительности. Он в автоматическом режиме ищет миллионы спектров у тысяч микробных геномов. Кроме того, NRPminer самостоятельно сравнивает полученные результаты с онлайн-базами данных и выделяет подходящие NRP. Исследователи уже используют этот инструмент для исследований и надеются обнаружить больше новых соединений для лекарственных препаратов. Материалы исследования опубликованы в статье «Integrating genomics and metabolomics for scalable non-ribosomal peptide discovery» в журнале Nature Doi.org/10.1038/s41467-021-23502-4.
===========
Источник:
habr.com
===========
Похожие новости:
- [Машинное обучение, Здоровье, Natural Language Processing] Тематическое исследование распознавания именованных сущностей в биомедицине (перевод)
- [История IT, Искусственный интеллект, Будущее здесь] Как открывали глаза компьютерам
- [Научно-популярное, Биотехнологии, Физика, Химия] Ученые создали подводный клей, наблюдая за моллюсками
- [Научно-популярное] Граф Цеппелин, открывший эпоху дирижаблей
- [Научно-популярное, Космонавтика] Следует ли Индии присоединиться к Китаю и российской лунной исследовательской станции?
- [Научно-популярное, Космонавтика] Dragon 2 с грузом на МКС. Запуски года: 50 всего, 24 от США
- [Open source, GitHub, Разработка под Windows, Софт, IT-компании] Microsoft вводит ручную модерацию пакетов проекта winget
- [Визуализация данных, Научно-популярное, Мозг, Здоровье, IT-компании] Google и Гарвард выпустили визуализацию коннектома человеческого мозга на 1,4 петабайта
- [Развитие стартапа, Научно-популярное, DIY или Сделай сам, Транспорт] Возрождение гражданской сверхзвуковой авиации
- [Искусственный интеллект] Умная одежда заменит термометр, флешку и не только
Теги для поиска: #_mashinnoe_obuchenie (Машинное обучение), #_nauchnopopuljarnoe (Научно-популярное), #_iskusstvennyj_intellekt (Искусственный интеллект), #_zdorove (Здоровье), #_meditsina (медицина), #_bakterii (бактерии), #_nejroseti (нейросети), #_mikrobiologija (микробиология), #_biohimija (биохимия), #_github, #_genetika (генетика), #_massspektrometrija (масс-спектрометрия), #_massspektrometr (масс-спектрометр), #_algoritmy (алгоритмы), #_mashinnoe_obuchenie (
Машинное обучение
), #_nauchnopopuljarnoe (
Научно-популярное
), #_iskusstvennyj_intellekt (
Искусственный интеллект
), #_zdorove (
Здоровье
)
Вы не можете начинать темы
Вы не можете отвечать на сообщения
Вы не можете редактировать свои сообщения
Вы не можете удалять свои сообщения
Вы не можете голосовать в опросах
Вы не можете прикреплять файлы к сообщениям
Вы не можете скачивать файлы
Текущее время: 11-Фев 12:24
Часовой пояс: UTC + 5
| Автор | Сообщение |
|---|---|
|
news_bot ®
Стаж: 8 лет |
|
|
Исследователи Университета Карнеги — Меллона обучили нейросеть искать в микробах метаболиты (нерибосомные пептиды) и оценивать их потенциальную пользу для разработки лекарств.  В процессе жизнедеятельности бактерии и грибки синтезируют вторичные метаболиты, в том числе нерибосомные пептиды (NRP). Эти пептиды учёные используют для производства антибиотиков, противоопухолевых препаратов и иммунодепрессантов. Например, один из самых мощных антибиотиков ванкомицин исследователи нашли в продуктах метаболизма бактерии Amycolatopsis orientalis. В последние годы исследователи отходят от поиска NRP в микробах. Процесс поиска трудоёмкий и требует много времени на оценку обнаруженных соединений. Не все пептиды пригодны для производства лекарств. Поэтому специалисты Университета Карнеги-Меллона создали алгоритм, упрощающий процессы поиска и анализа. Исследователи назвали платформу NRPminer и выложили на GitHub. Для исследования учёные взяли метаболиты штаммов микробов семейств Actinobacteria, Xenorhabdus, Photorhabdus, Staphylococcus и почвенных бактерий Bacillus, Pseudomonas, Buttiauxella и Rahnella. Всего отобрали 246 штаммов. В ходе анализа NRPminer идентифицировала биосинтетические генные кластеры пептидов (BGC), используя пайплайн antiSMASH16. (пункт “а” на рисунке ниже) Система предсказывала вариации аминокислот с помощью веб-сервера NRPSpredictor2. (b) Далее она рассмотрела сформированные NRPS последовательности нуклеотидов. (с) NRPminer отфильтровала набор основных последовательностей NRP на основе оценки специфичности аминокислот и выбрала варианты с самыми высокими оценками. (d) Платформа нашла каждый BGC и включила их в последовательности NRP. (e) Она создала базу данных потенциальных структур NPR (f), после чего искала подходящие модификации и вычислила их статистическую значимость. (g) Компонент NRPS выделил и расширил статистически значимые показатели с использованием метода спектральных сетей. (h и i) Обнаруженные вариации NRP алгоритм сопоставил с общедоступным набором спектральных данных геномов RefSeq82 и онлайн-базой данных геномов GOLD.  В итоге NRPminer обнаружил 13 известных семейств NRP (55 вариаций) и 4 неизвестных семейства с 25 вариациями. Исследователи уже изучили неизвестные семейства и выделили у двух антипаразитарную активность. Кроме того, анализ показал наличие 121 известной вариации NRP у микробов, к которым исследователи ранее не относили эти пептиды. Алгоритм определяет только 1 % продуктов метаболизма микробов. Остальные 99 % включают в себя первичные метаболиты (аминокислоты), другие вторичные метаболиты (RiPP, поликетиды, липиды, терпены), молекулы из окружающей среды и элементы с низкой интенсивностью, которые сложно обнаружить.  Спектральные сети для девяти известных и трёх ранее неизвестных семейств NRP. Каждый узел на изображении обозначает спектр NRP. Узел окрашен, если спектр обладает статистической значимостью. Известные NRP окрашены голубым. Светло-зелёным отмечены неизвестные NRP. Спектры ранее неизвестных семейств NRP окрашены сиреневым.Исследователи отмечают необходимость дальнейшего пополнения базы данных и повышения чувствительности NRPminer. В ходе анализа авторы столкнулись с проблемой наличия в результатах масс-спектрометрии спектров из окружающей среды. Например, большинство спектров микробиома кожи человека соответствует косметических средствам (крема, дезодоранты). Учёные заявили, что появление более чувствительных методом спектрометрии решит эту проблему. Согласно заявлению авторов исследования, NRPminer — первый генетический инструмент, эффективно фильтрующий основные NRP на основе показателей специфичности без потери чувствительности. Он в автоматическом режиме ищет миллионы спектров у тысяч микробных геномов. Кроме того, NRPminer самостоятельно сравнивает полученные результаты с онлайн-базами данных и выделяет подходящие NRP. Исследователи уже используют этот инструмент для исследований и надеются обнаружить больше новых соединений для лекарственных препаратов. Материалы исследования опубликованы в статье «Integrating genomics and metabolomics for scalable non-ribosomal peptide discovery» в журнале Nature Doi.org/10.1038/s41467-021-23502-4. =========== Источник: habr.com =========== Похожие новости:
Машинное обучение ), #_nauchnopopuljarnoe ( Научно-популярное ), #_iskusstvennyj_intellekt ( Искусственный интеллект ), #_zdorove ( Здоровье ) |
|
Вы не можете начинать темы
Вы не можете отвечать на сообщения
Вы не можете редактировать свои сообщения
Вы не можете удалять свои сообщения
Вы не можете голосовать в опросах
Вы не можете прикреплять файлы к сообщениям
Вы не можете скачивать файлы
Вы не можете отвечать на сообщения
Вы не можете редактировать свои сообщения
Вы не можете удалять свои сообщения
Вы не можете голосовать в опросах
Вы не можете прикреплять файлы к сообщениям
Вы не можете скачивать файлы
Текущее время: 11-Фев 12:24
Часовой пояс: UTC + 5
