[Алгоритмы, Обработка изображений, Машинное обучение] Генетический алгоритм для сегментаций строк в рукописном документе
Автор
Сообщение
news_bot ®
Стаж: 7 лет 10 месяцев
Сообщений: 27286
Генетический алгоритм (GA)Генетический алгоритм - это классический эволюционный алгоритм, основанный на случайной переборе параметр. Под случайным здесь мы подразумеваем, что для поиска решения с использованием ГА, случайные изменения применялись к текущим решениям для генерации новых. GA основан на теории эволюции Дарвина. Это медленный постепенный процесс, который работает путем внесения небольших и медленных изменений. Кроме того, GA медленно вносит небольшие изменения в свои решения, пока не получит лучшее решение. Подробнее можете узнать здесьЦельРеализовать алгоритм для сегментаций строк в рукописном документе(рис. 1), чтобы при обрезаний строк, не обрезали нижнюю или верхнюю часть букв (Например: рукописные буквы р, в, б, д, з и т.д.).
Рис. 1 ОбразецРеализацияPreprocessing Для начала, нужно узнать диапазон случайных чисел, где мы будем рисовать линию(индивид). Для этого, построил гистограмму картинки(рис. 2. По сумме цветов каждого пикселя, например: если строка полностью белая, то сумма пикселей будет равна 255) по высоте картинки. Чтобы было удобно, можно сделать инверсию, тогда белый цвет будет равен нулю.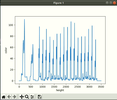
Рис. 2Это гистограмма дает нам сумму пикселей каждой строки изображения, как видите слишком много пиков. Теперь нам надо сглаживать(рис. 3) эту гистограмму с помощью фильтра гаусса(есть и другие фильтры). Так мы получим точные координаты пикселей, где преобладает черный или белый цвет.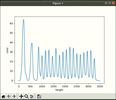
Рис. 3Если рисовать линии по максимальным(синяя) и минимальным(зеленая) точкам по этой гистограмме, то получится нижняя картинка(рис. 4)
Рис. 4Можно считать, что зеленая линия разделяет строки, но как видим, они пересекают верхнюю или нижнюю часть некоторых букв, именно поэтому, мы использовали генетический алгоритм, чтобы обходить такие буквы. Синие линии будут служить нам, как диапазон для индивидов, именно в этом диапазоне мы будем рисовать линии.Генетический алгоритмДля начала, создадим интерфейс нашего алгоритма, так мы можем использовать свойство полиморфизм:
from abc import abstractmethod, abstractproperty
class GeneticAlgorithm():
"""Genetic Algorithm Interface."""
@abstractmethod
def create_population(self):
pass
@abstractmethod
def selection(self):
pass
@abstractmethod
def mutation(self):
pass
@abstractmethod
def cross(self):
pass
@abstractmethod
def call(self):
pass
Интерфейс состоит из таких функций:
- create_population - создание начальной популяций;
- selection - алгоритм отбора;
- mutation - алгоритм мутации;
- cross - алгоритм скрещивания;
- call - функция, которая вызывает предедущие 3 функций;
Прежде чем приступать к реализаций алгоритма, нам нужно создать индивида. Индивид объявлен следующим образом:
import numpy as np
import random
class Individ:
"""Generate a new individ."""
def __init__(self, y1, y2, length):
self.length = length
self.A=np.random.randint(y1, y2, self.length) # Declare the attribute "A" of our individ (this will be the line 010..101).
self.fit=0 # Declare the attribute "fit" of the individ (this will be the sum of the elements of our line 010..101) (for now, we will assign it the value 0)
Класс Individ должен возвращать вектор хромосом, на основе которых будет создана начальная популяция.Теперь можно реализовать алгоритм, используя наследование от этого абстрактного класса. В конструктор для инициализаций, передаем количество популяции, количество индивидов, длину индивидов в пикселях и картинку в серых тонах.
class SimpleSegmentationGA(GeneticAlgorithm):
"""Genetic Algorithm Implementation."""
def __init__(self, pop_num, lenght, delta_x, gray):
self.pop_num = pop_num
self.lenght = lenght # Length of individ.
self.delta_x = delta_x # Value of h.
self.gray = 256 - gray # Inversion.
Функция create_population описывает создание начальной популяции.
def create_population(self, y1, y2):
"""Creates a population, Dad, Mom and 4 sons"""
self.y1 = y1
self.y2 = y2
self.population = [] # Declare an array "population" in which we will store a population of 5 individuals.
for i in range(self.pop_num):
c=Individ(self.y1, self.y2, self.lenght) # Create a new individual.
self.population.append(c) # Add the i-th unique individual to the array (fill in our population)
self.mother = Individ(self.y1, self.y2, self.lenght) # Initialize the variables with which we will work: mom, dad, 4 sons ..
self.father = Individ(self.y1, self.y2, self.lenght)
self.son1 = Individ(self.y1, self.y2, self.lenght)
self.son2 = Individ(self.y1, self.y2, self.lenght)
self.son3 = Individ(self.y1, self.y2, self.lenght)
self.son4 = Individ(self.y1, self.y2, self.lenght)
self.par_and_sons = [] #.. and an array of individs "Parents and Children" in which we will store
for j in range(self.pop_num*3): # Initialize our array of "Parents and Sons" with random individs.
self.par_and_sons.append(Individ(self.y1, self.y2, self.lenght))
Функция create_population создает родителей, а также 4 сыновей. После того, как были сформированы «семьи» из особей, для скрещивания, нужно скрестить их хромосомы, чтобы получить детей с новыми хромосомами. Функция cross описывает скрещивание следующим образом:
def cross(self):
"""Crosses the best individs with each other."""
for i in range(self.pop_num): # Put in the first pop_num elements of the "Parents and Sons" array our entire input population.
self.par_and_sons[i].A=self.population[i].A.copy()
random.shuffle(self.population) # Shuffle population.
tt=0 # The counter that is needed to implement a non-trivial crossing.
for s in range(0,self.pop_num,2): # From 0 to pop_num with step 2. That is. here we take pop_num / 2 pairs of parents.
self.mother.A=self.population[tt+int(self.pop_num/2)].A # Let the last pop_num / 2 individuals of our population be our mothers.
self.father.A=self.population[tt].A # And let first pop_num / 2 individuals of our population be dads.
tt=tt+1
ran=random.random()
for n in range(self.lenght): # Crossover.
if random.random()>0.5:
self.son1.A[n] = self.father.A[n]
self.son2.A[self.lenght-1-n] = self.father.A[n]
self.son3.A[n] = self.mother.A[n]
self.son4.A[self.lenght-1-n] = self.mother.A[n]
else:
self.son1.A[n] = self.mother.A[n]
self.son2.A[self.lenght-1-n] = self.mother.A[n]
self.son3.A[n] = self.father.A[n]
self.son4.A[self.lenght-1-n] = self.father.A[n]
self.par_and_sons[self.pop_num+2*s].A = self.son1.A.copy()
self.par_and_sons[self.pop_num+2*s+1].A = self.son2.A.copy()
self.par_and_sons[self.pop_num+2*s+2].A = self.son3.A.copy()
self.par_and_sons[self.pop_num+2*s+3].A = self.son4.A.copy()
После скрещивания необходимо провести мутацию полученных детей, чтобы поддерживать разнообразие хромосом, и популяция не скатилась к вырожденному состоянию. Метод mutation объявлен следующим образом:
def mutation(self):
"""Mutates individuals with a 20% probability."""
for r in range(self.pop_num*3, 5): # Mutation.
for w in range(0,self.lenght):
if random.random()<0.2:
self.par_and_sons[r].A[w] = self.par_and_sons[r].A[w] + np.random.randint(-20, 20) # Offset + -20 pixels.
Метод mutation должен изменить хромосому, которая поступает к нему. Как правило, устанавливают небольшую вероятность мутации, например, единицы процентов, чтобы все-таки сохранялись хромосомы, полученные на основе родителей и таким образом улучшали популяцию.После скрещивания и мутации обычно наступает этап отбора, на котором уничтожаются самые неудачные особи. Для этого используется метод selection, который объявлен следующим образом:
def selection(self):
"""Sorts by fit and selects the best pop_num individs."""
for i in range(self.pop_num*3): # It is important. Next, we will rank the array of parents and children in ascending order of survivability (sum (fit)).
self.par_and_sons[i].fit = SimpleSegmentationGA.fitness_function(self.gray, self.delta_x, self.lenght, self.par_and_sons[i].A)
# Sort.
self.par_and_sons = sorted(self.par_and_sons, key=lambda individ: individ.fit)
self.population=self.par_and_sons[:self.pop_num].copy()
В методе происходить расчет фитнес функций и сортировка по нему. По окончанию сортировки берется первые pop_num элементов, так как они являются лучшими из этого поколения.Фитнес функция объявлен следующим образом:
def fitness(image, delta_x, length, individ):
"""Responsible for calculating the "fit" (counts the amount)."""
summa = 0
sum_vrt = 0
for i in range(length):
sum_ = np.sum(image[individ[i], i*delta_x:i*delta_x+delta_x])
if i>0:
if individ[i]>individ[i-1]:
sum_vrt = np.sum(image[individ[i-1]:individ[i], i*delta_x])
else:
sum_vrt = np.sum(image[individ[i]:individ[i-1], i*delta_x])
summa=summa + sum_ + sum_vrt
return summa
В этой функций мы учитываем сумму горизонтальных, а также соединяющих линии. У нас белый цвет имеет вес 1, а черный 256.Последний метод call который вызывает три других метода объявлен следующим образом:
def call(self):
"""Calls other functions and returns the selected population."""
self.cross()
self.mutation()
self.selection()
return self.population[0]
Метод call должен возвращать отобранную популяцию.Также были реализованы вспомагательные функций для нахождения динапазона для индивидов, для рисования результатов, для обрезки результатов, а также для запуска генетического алгоритма.
import numpy as np
import random, time
import cv2
from scipy.ndimage import gaussian_filter
from scipy.signal import find_peaks
from numba import njit
import pymp
@njit
def fitness(image, delta_x, length, individ):
"""Responsible for calculating the "fit" (counts the amount)."""
summa = 0
sum_vrt = 0
for i in range(length):
sum_ = np.sum(image[individ[i], i*delta_x:i*delta_x+delta_x])
if i>0:
if individ[i]>individ[i-1]:
sum_vrt = np.sum(image[individ[i-1]:individ[i], i*delta_x])
else:
sum_vrt = np.sum(image[individ[i]:individ[i-1], i*delta_x])
summa=summa + sum_ + sum_vrt
return summa
def find_peaks_(image):
"""Calculates ranges of random numbers for our individs."""
height, width = image.shape[:2]
img_matrix = [sum(i)/len(i) for i in image]
x=[i for i in range(height)]
y = [255-i for i in img_matrix]
y = gaussian_filter(y, sigma=20)
maxs, _ = find_peaks(y)
maxs = maxs.tolist()
return maxs
def run(gaObj, peaks, epoch, parallel=True, number_cpu=4):
"""Can choose how to run."""
gen_lines = pymp.shared.list()
if parallel:
with pymp.Parallel(number_cpu) as p:
range_peaks = p.range(len(peaks)-1)
gen_lines = run_genetic_algorithm(gaObj, peaks, epoch, range_peaks, gen_lines)
else:
range_peaks = range(len(peaks)-1)
gen_lines = run_genetic_algorithm(gaObj, peaks, epoch, range_peaks, gen_lines)
return np.moveaxis(np.array(gen_lines), 0, 1) # Swap line and epoch axes.
def draw_lines(image, lines, delta_x, imagename, epoch='last', color=(0,0,255), thickness = 3, IMG_FOLDER='output'):
"""Draws the result of a genetic algorithm."""
image_copy = image.copy()
for j in lines:
for i in range(len(j)):
x1 = i*delta_x
x2 = i*delta_x+delta_x
y1 = j[i]
y2 = j[i]
start_point = (x1, y1)
if i>0:
cv2.line(image_copy, preview_point, start_point, color, thickness)
end_point = (x2, y2)
cv2.line(image_copy, start_point, end_point, color, thickness)
preview_point = end_point
cv2.imwrite(f"{IMG_FOLDER}/{imagename[:-4]}_gen_line_{epoch}.jpg", image_copy)
def run_genetic_algorithm(gaObj, peaks, epoch, range_peaks, gen_lines):
"""Runs a genetic alghoritm."""
for line in range_peaks:
gaObj.create_population(peaks[line], peaks[line+1])
epoch_line = pymp.shared.list()
for p in range(epoch):
st_time = time.time()
gen_line = gaObj.call()
epoch_line.append(gen_line.A) # For draw results each epoch, add results of each epoch.
print(f'Line = {line}, Epoch = {p}, fit = {gen_line.fit}, Time = {time.time()-st_time}')
gen_lines.append(epoch_line)
return gen_lines
def crop_lines(image, lines, delta_x, imagename, IMG_FOLDER='crops'):
"""Crop the lines."""
for i in range(-1,len(lines)):
height, width = image.shape[:2]
if i == (-1):
first_line = [[i*delta_x, 0] for i, el in enumerate(lines[i])]
else:
first_line = [[i*delta_x, el] for i, el in enumerate(lines[i])]
first_line = first_line[::-1] # Reverse first line.
if i < len(lines)-1:
second_line = [[i*delta_x, el] for i, el in enumerate(lines[i+1])]
else:
second_line = [[i*delta_x, height] for i, el in enumerate(lines[i])]
points = np.array([first_line + second_line])
mask = np.zeros((height, width), dtype=np.uint8)
cv2.fillPoly(mask, points, (255))
res = cv2.bitwise_and(image,image,mask = mask)
rect = cv2.boundingRect(points) # Returns (x,y,w,h) of the rect
im2 = np.full((res.shape[0], res.shape[1], 3), (0, 255, 0), dtype=np.uint8 ) # You can also use other colors or simply load another image of the same size
maskInv = cv2.bitwise_not(mask)
colorCrop = cv2.bitwise_or(im2,im2,mask = maskInv)
finalIm = res + colorCrop
cropped = finalIm[rect[1]: rect[1] + rect[3], rect[0]: rect[0] + rect[2]]
cv2.imwrite(f"{IMG_FOLDER}/croped_{imagename[:-4]}_{i}.png", cropped)
Так как, генетический алгоритм является итерационной задачей, он занимает много времени, основное время занимает расчет фитнес функций. Чтобы увеличить время обработки мы использовали библиотеку numba для фитнес функций и уменшили скорость обработки в 27 раз(190sec VS 7sec для одного изображения). Также использовали библиотеку pymp для распараллеливания, итого время сократилась до 3,5 сек. на одно изображение.Запускаемый файл объявлен следующим образом:
import cv2
import glob, os
from utils import *
from SimpleSegmentationGA import SimpleSegmentationGA
if __name__ == '__main__':
# Hyper parameters for genetic alghoritms.
POP_NUM=80
DELTAX=50 # Value of h.
EPOCH=100
IMAGES_PATH = 'input'
# IMAGE_PATH = '7.jpg'
images = glob.glob(f'{IMAGES_PATH}/*.jpg')
for imagename in images:
fstart_time = time.time()
# Image operations.
image = cv2.imread(imagename)
height, width = image.shape[:2]
gray = cv2.cvtColor(image, cv2.COLOR_BGR2GRAY)
# Calculate individ's lenght.
length_individ = int(width/DELTAX)
# Calculate the range of random numbers for an individ.
peaks = find_peaks_(gray)
# Create object of class.
ssga = SimpleSegmentationGA(POP_NUM, length_individ, DELTAX, gray)
start_time = time.time()
# Run genetic algorithm.
lines = run(ssga, peaks, EPOCH, parallel=True)
print('Time run =', time.time()-start_time)
last_epoch_lines = sorted(lines[-1], key = lambda x:x[0]) # Sort for crop. Because when using parallel, the lines lose consistency.
start_time = time.time()
# Draw the result of the genetic alghoritm. lines[-1] -> result of last epoch.
draw_lines(image, last_epoch_lines, DELTAX, os.path.basename(imagename))
print('Time draw_lines =', time.time()-start_time)
start_time = time.time()
# Crop the result of genetic alghoritm.
crop_lines(image, last_epoch_lines, DELTAX, os.path.basename(imagename))
print('Time crop_lines =', time.time()-start_time)
print('Time for one image =', time.time()-fstart_time)
Результат оправдал ожидание:
Рис. 5Исходный код можете найти здесь.
===========
Источник:
habr.com
===========
Похожие новости:
- [Программирование, Машинное обучение] Искусственный интеллект обновит устаревшее программное обеспечение за вас (перевод)
- [Python, Машинное обучение, Искусственный интеллект, TensorFlow] Нейродайджест: главное из области машинного обучения за февраль 2021
- [Машинное обучение] Как главная страница ivi оптимизируется под пользователей и их устройства?
- [Обработка изображений, Хранение данных, Хранилища данных] На Google Photos начнут сжимать качественные фото с искажениями
- [Python, Алгоритмы, Карьера в IT-индустрии, Natural Language Processing] Как улучшить резюме с помощью алгоритмов обработки текстов на естественных языках (перевод)
- [Обработка изображений, Научно-популярное, Космонавтика] Ученые объяснили, как камеры «Персеверанса» видят Марс
- [Машинное обучение, Искусственный интеллект, IT-компании, Natural Language Processing] DeepPavlov 3 года: обзор и итоги 2020 года
- [Машинное обучение, Искусственный интеллект, Natural Language Processing] NLP (Natural Language Processing) для обращений граждан. Эксперимент на реальных данных
- [Data Mining, Машинное обучение, Искусственный интеллект, Финансы в IT, Data Engineering] Специфические задачи Data Science в Банке
- [Алгоритмы, Читальный зал, Искусственный интеллект, Логические игры] Шахматные алгоритмы, которые думают почти так же, как человек, только лучше
Теги для поиска: #_algoritmy (Алгоритмы), #_obrabotka_izobrazhenij (Обработка изображений), #_mashinnoe_obuchenie (Машинное обучение), #_geneticheskij_algoritm (генетический алгоритм), #_obrabotka_izobrazhenij (обработка изображений), #_algoritmy (
Алгоритмы
), #_obrabotka_izobrazhenij (
Обработка изображений
), #_mashinnoe_obuchenie (
Машинное обучение
)
Вы не можете начинать темы
Вы не можете отвечать на сообщения
Вы не можете редактировать свои сообщения
Вы не можете удалять свои сообщения
Вы не можете голосовать в опросах
Вы не можете прикреплять файлы к сообщениям
Вы не можете скачивать файлы
Текущее время: 27-Дек 04:29
Часовой пояс: UTC + 5
| Автор | Сообщение |
|---|---|
|
news_bot ®
Стаж: 7 лет 10 месяцев |
|
|
Генетический алгоритм (GA)Генетический алгоритм - это классический эволюционный алгоритм, основанный на случайной переборе параметр. Под случайным здесь мы подразумеваем, что для поиска решения с использованием ГА, случайные изменения применялись к текущим решениям для генерации новых. GA основан на теории эволюции Дарвина. Это медленный постепенный процесс, который работает путем внесения небольших и медленных изменений. Кроме того, GA медленно вносит небольшие изменения в свои решения, пока не получит лучшее решение. Подробнее можете узнать здесьЦельРеализовать алгоритм для сегментаций строк в рукописном документе(рис. 1), чтобы при обрезаний строк, не обрезали нижнюю или верхнюю часть букв (Например: рукописные буквы р, в, б, д, з и т.д.).  Рис. 1 ОбразецРеализацияPreprocessing Для начала, нужно узнать диапазон случайных чисел, где мы будем рисовать линию(индивид). Для этого, построил гистограмму картинки(рис. 2. По сумме цветов каждого пикселя, например: если строка полностью белая, то сумма пикселей будет равна 255) по высоте картинки. Чтобы было удобно, можно сделать инверсию, тогда белый цвет будет равен нулю.  Рис. 2Это гистограмма дает нам сумму пикселей каждой строки изображения, как видите слишком много пиков. Теперь нам надо сглаживать(рис. 3) эту гистограмму с помощью фильтра гаусса(есть и другие фильтры). Так мы получим точные координаты пикселей, где преобладает черный или белый цвет.  Рис. 3Если рисовать линии по максимальным(синяя) и минимальным(зеленая) точкам по этой гистограмме, то получится нижняя картинка(рис. 4)  Рис. 4Можно считать, что зеленая линия разделяет строки, но как видим, они пересекают верхнюю или нижнюю часть некоторых букв, именно поэтому, мы использовали генетический алгоритм, чтобы обходить такие буквы. Синие линии будут служить нам, как диапазон для индивидов, именно в этом диапазоне мы будем рисовать линии.Генетический алгоритмДля начала, создадим интерфейс нашего алгоритма, так мы можем использовать свойство полиморфизм: from abc import abstractmethod, abstractproperty
class GeneticAlgorithm(): """Genetic Algorithm Interface.""" @abstractmethod def create_population(self): pass @abstractmethod def selection(self): pass @abstractmethod def mutation(self): pass @abstractmethod def cross(self): pass @abstractmethod def call(self): pass
import numpy as np
import random class Individ: """Generate a new individ.""" def __init__(self, y1, y2, length): self.length = length self.A=np.random.randint(y1, y2, self.length) # Declare the attribute "A" of our individ (this will be the line 010..101). self.fit=0 # Declare the attribute "fit" of the individ (this will be the sum of the elements of our line 010..101) (for now, we will assign it the value 0) class SimpleSegmentationGA(GeneticAlgorithm):
"""Genetic Algorithm Implementation.""" def __init__(self, pop_num, lenght, delta_x, gray): self.pop_num = pop_num self.lenght = lenght # Length of individ. self.delta_x = delta_x # Value of h. self.gray = 256 - gray # Inversion. def create_population(self, y1, y2):
"""Creates a population, Dad, Mom and 4 sons""" self.y1 = y1 self.y2 = y2 self.population = [] # Declare an array "population" in which we will store a population of 5 individuals. for i in range(self.pop_num): c=Individ(self.y1, self.y2, self.lenght) # Create a new individual. self.population.append(c) # Add the i-th unique individual to the array (fill in our population) self.mother = Individ(self.y1, self.y2, self.lenght) # Initialize the variables with which we will work: mom, dad, 4 sons .. self.father = Individ(self.y1, self.y2, self.lenght) self.son1 = Individ(self.y1, self.y2, self.lenght) self.son2 = Individ(self.y1, self.y2, self.lenght) self.son3 = Individ(self.y1, self.y2, self.lenght) self.son4 = Individ(self.y1, self.y2, self.lenght) self.par_and_sons = [] #.. and an array of individs "Parents and Children" in which we will store for j in range(self.pop_num*3): # Initialize our array of "Parents and Sons" with random individs. self.par_and_sons.append(Individ(self.y1, self.y2, self.lenght)) def cross(self):
"""Crosses the best individs with each other.""" for i in range(self.pop_num): # Put in the first pop_num elements of the "Parents and Sons" array our entire input population. self.par_and_sons[i].A=self.population[i].A.copy() random.shuffle(self.population) # Shuffle population. tt=0 # The counter that is needed to implement a non-trivial crossing. for s in range(0,self.pop_num,2): # From 0 to pop_num with step 2. That is. here we take pop_num / 2 pairs of parents. self.mother.A=self.population[tt+int(self.pop_num/2)].A # Let the last pop_num / 2 individuals of our population be our mothers. self.father.A=self.population[tt].A # And let first pop_num / 2 individuals of our population be dads. tt=tt+1 ran=random.random() for n in range(self.lenght): # Crossover. if random.random()>0.5: self.son1.A[n] = self.father.A[n] self.son2.A[self.lenght-1-n] = self.father.A[n] self.son3.A[n] = self.mother.A[n] self.son4.A[self.lenght-1-n] = self.mother.A[n] else: self.son1.A[n] = self.mother.A[n] self.son2.A[self.lenght-1-n] = self.mother.A[n] self.son3.A[n] = self.father.A[n] self.son4.A[self.lenght-1-n] = self.father.A[n] self.par_and_sons[self.pop_num+2*s].A = self.son1.A.copy() self.par_and_sons[self.pop_num+2*s+1].A = self.son2.A.copy() self.par_and_sons[self.pop_num+2*s+2].A = self.son3.A.copy() self.par_and_sons[self.pop_num+2*s+3].A = self.son4.A.copy() def mutation(self):
"""Mutates individuals with a 20% probability.""" for r in range(self.pop_num*3, 5): # Mutation. for w in range(0,self.lenght): if random.random()<0.2: self.par_and_sons[r].A[w] = self.par_and_sons[r].A[w] + np.random.randint(-20, 20) # Offset + -20 pixels. def selection(self):
"""Sorts by fit and selects the best pop_num individs.""" for i in range(self.pop_num*3): # It is important. Next, we will rank the array of parents and children in ascending order of survivability (sum (fit)). self.par_and_sons[i].fit = SimpleSegmentationGA.fitness_function(self.gray, self.delta_x, self.lenght, self.par_and_sons[i].A) # Sort. self.par_and_sons = sorted(self.par_and_sons, key=lambda individ: individ.fit) self.population=self.par_and_sons[:self.pop_num].copy() def fitness(image, delta_x, length, individ):
"""Responsible for calculating the "fit" (counts the amount).""" summa = 0 sum_vrt = 0 for i in range(length): sum_ = np.sum(image[individ[i], i*delta_x:i*delta_x+delta_x]) if i>0: if individ[i]>individ[i-1]: sum_vrt = np.sum(image[individ[i-1]:individ[i], i*delta_x]) else: sum_vrt = np.sum(image[individ[i]:individ[i-1], i*delta_x]) summa=summa + sum_ + sum_vrt return summa def call(self):
"""Calls other functions and returns the selected population.""" self.cross() self.mutation() self.selection() return self.population[0] import numpy as np
import random, time import cv2 from scipy.ndimage import gaussian_filter from scipy.signal import find_peaks from numba import njit import pymp @njit def fitness(image, delta_x, length, individ): """Responsible for calculating the "fit" (counts the amount).""" summa = 0 sum_vrt = 0 for i in range(length): sum_ = np.sum(image[individ[i], i*delta_x:i*delta_x+delta_x]) if i>0: if individ[i]>individ[i-1]: sum_vrt = np.sum(image[individ[i-1]:individ[i], i*delta_x]) else: sum_vrt = np.sum(image[individ[i]:individ[i-1], i*delta_x]) summa=summa + sum_ + sum_vrt return summa def find_peaks_(image): """Calculates ranges of random numbers for our individs.""" height, width = image.shape[:2] img_matrix = [sum(i)/len(i) for i in image] x=[i for i in range(height)] y = [255-i for i in img_matrix] y = gaussian_filter(y, sigma=20) maxs, _ = find_peaks(y) maxs = maxs.tolist() return maxs def run(gaObj, peaks, epoch, parallel=True, number_cpu=4): """Can choose how to run.""" gen_lines = pymp.shared.list() if parallel: with pymp.Parallel(number_cpu) as p: range_peaks = p.range(len(peaks)-1) gen_lines = run_genetic_algorithm(gaObj, peaks, epoch, range_peaks, gen_lines) else: range_peaks = range(len(peaks)-1) gen_lines = run_genetic_algorithm(gaObj, peaks, epoch, range_peaks, gen_lines) return np.moveaxis(np.array(gen_lines), 0, 1) # Swap line and epoch axes. def draw_lines(image, lines, delta_x, imagename, epoch='last', color=(0,0,255), thickness = 3, IMG_FOLDER='output'): """Draws the result of a genetic algorithm.""" image_copy = image.copy() for j in lines: for i in range(len(j)): x1 = i*delta_x x2 = i*delta_x+delta_x y1 = j[i] y2 = j[i] start_point = (x1, y1) if i>0: cv2.line(image_copy, preview_point, start_point, color, thickness) end_point = (x2, y2) cv2.line(image_copy, start_point, end_point, color, thickness) preview_point = end_point cv2.imwrite(f"{IMG_FOLDER}/{imagename[:-4]}_gen_line_{epoch}.jpg", image_copy) def run_genetic_algorithm(gaObj, peaks, epoch, range_peaks, gen_lines): """Runs a genetic alghoritm.""" for line in range_peaks: gaObj.create_population(peaks[line], peaks[line+1]) epoch_line = pymp.shared.list() for p in range(epoch): st_time = time.time() gen_line = gaObj.call() epoch_line.append(gen_line.A) # For draw results each epoch, add results of each epoch. print(f'Line = {line}, Epoch = {p}, fit = {gen_line.fit}, Time = {time.time()-st_time}') gen_lines.append(epoch_line) return gen_lines def crop_lines(image, lines, delta_x, imagename, IMG_FOLDER='crops'): """Crop the lines.""" for i in range(-1,len(lines)): height, width = image.shape[:2] if i == (-1): first_line = [[i*delta_x, 0] for i, el in enumerate(lines[i])] else: first_line = [[i*delta_x, el] for i, el in enumerate(lines[i])] first_line = first_line[::-1] # Reverse first line. if i < len(lines)-1: second_line = [[i*delta_x, el] for i, el in enumerate(lines[i+1])] else: second_line = [[i*delta_x, height] for i, el in enumerate(lines[i])] points = np.array([first_line + second_line]) mask = np.zeros((height, width), dtype=np.uint8) cv2.fillPoly(mask, points, (255)) res = cv2.bitwise_and(image,image,mask = mask) rect = cv2.boundingRect(points) # Returns (x,y,w,h) of the rect im2 = np.full((res.shape[0], res.shape[1], 3), (0, 255, 0), dtype=np.uint8 ) # You can also use other colors or simply load another image of the same size maskInv = cv2.bitwise_not(mask) colorCrop = cv2.bitwise_or(im2,im2,mask = maskInv) finalIm = res + colorCrop cropped = finalIm[rect[1]: rect[1] + rect[3], rect[0]: rect[0] + rect[2]] cv2.imwrite(f"{IMG_FOLDER}/croped_{imagename[:-4]}_{i}.png", cropped) import cv2
import glob, os from utils import * from SimpleSegmentationGA import SimpleSegmentationGA if __name__ == '__main__': # Hyper parameters for genetic alghoritms. POP_NUM=80 DELTAX=50 # Value of h. EPOCH=100 IMAGES_PATH = 'input' # IMAGE_PATH = '7.jpg' images = glob.glob(f'{IMAGES_PATH}/*.jpg') for imagename in images: fstart_time = time.time() # Image operations. image = cv2.imread(imagename) height, width = image.shape[:2] gray = cv2.cvtColor(image, cv2.COLOR_BGR2GRAY) # Calculate individ's lenght. length_individ = int(width/DELTAX) # Calculate the range of random numbers for an individ. peaks = find_peaks_(gray) # Create object of class. ssga = SimpleSegmentationGA(POP_NUM, length_individ, DELTAX, gray) start_time = time.time() # Run genetic algorithm. lines = run(ssga, peaks, EPOCH, parallel=True) print('Time run =', time.time()-start_time) last_epoch_lines = sorted(lines[-1], key = lambda x:x[0]) # Sort for crop. Because when using parallel, the lines lose consistency. start_time = time.time() # Draw the result of the genetic alghoritm. lines[-1] -> result of last epoch. draw_lines(image, last_epoch_lines, DELTAX, os.path.basename(imagename)) print('Time draw_lines =', time.time()-start_time) start_time = time.time() # Crop the result of genetic alghoritm. crop_lines(image, last_epoch_lines, DELTAX, os.path.basename(imagename)) print('Time crop_lines =', time.time()-start_time) print('Time for one image =', time.time()-fstart_time)  Рис. 5Исходный код можете найти здесь. =========== Источник: habr.com =========== Похожие новости:
Алгоритмы ), #_obrabotka_izobrazhenij ( Обработка изображений ), #_mashinnoe_obuchenie ( Машинное обучение ) |
|
Вы не можете начинать темы
Вы не можете отвечать на сообщения
Вы не можете редактировать свои сообщения
Вы не можете удалять свои сообщения
Вы не можете голосовать в опросах
Вы не можете прикреплять файлы к сообщениям
Вы не можете скачивать файлы
Вы не можете отвечать на сообщения
Вы не можете редактировать свои сообщения
Вы не можете удалять свои сообщения
Вы не можете голосовать в опросах
Вы не можете прикреплять файлы к сообщениям
Вы не можете скачивать файлы
Текущее время: 27-Дек 04:29
Часовой пояс: UTC + 5
